t-distributed Stochastic Neighbor Embedding (t-SNE)
Entdecken Sie, wie t-SNE hochdimensionale Daten visualisiert. Lernen Sie, Cluster in Computer-Vision-Features für Ultralytics aufzudecken und Machine-Learning-Modelle zu optimieren.
t-verteilte stochastische Nachbarschaftseinbettung (t-SNE) ist eine statistische Methode zur Visualisierung hochdimensionaler Daten, indem
jedem Datenpunkt eine Position in einer zwei- oder dreidimensionalen Karte zugewiesen wird. Diese Technik, eine Form der nichtlinearen
Dimensionsreduktion, wird häufig
im maschinellen Lernen eingesetzt, um Datensätze mit Hunderten oder Tausenden von Merkmalen zu untersuchen. Im Gegensatz zu linearen Methoden, die
sich auf die Erhaltung globaler Strukturen konzentrieren, ist t-SNE besonders gut darin, ähnliche Instanzen nahe beieinander zu halten und lokale
Cluster und Mannigfaltigkeiten aufzudecken, die sonst möglicherweise verborgen blieben. Dies macht es zu einem unschätzbaren Werkzeug für alles, von der
Genomforschung bis zum Verständnis der internen Logik tiefer neuronaler Netze.
Wie t-SNE funktioniert
Die Kernidee hinter t-SNE besteht darin, die Ähnlichkeiten zwischen Datenpunkten in gemeinsame Wahrscheinlichkeiten umzuwandeln. Im
ursprünglichen hochdimensionalen Raum misst der Algorithmus die Ähnlichkeit zwischen Punkten anhand einer Gaußschen Verteilung.
Wenn zwei Punkte nahe beieinander liegen, ist die Wahrscheinlichkeit hoch, dass sie „Nachbarn” sind. Der Algorithmus versucht dann,
diese Punkte in einen Raum mit geringerer Dimension (in der Regel 2D oder 3D) abzubilden und dabei diese Wahrscheinlichkeiten beizubehalten.
Um dies zu erreichen, definiert es eine ähnliche Wahrscheinlichkeitsverteilung in der niedrigdimensionalen Karte unter Verwendung einer Student-t-Verteilung
. Diese spezifische Verteilung hat schwerere Enden als eine normale Gauß-Verteilung, was dazu beiträgt, das
„Crowding-Problem” zu lösen – ein Phänomen, bei dem Punkte im hochdimensionalen Raum dazu neigen, sich bei der Projektion übereinander zu
lagern. Indem t-SNE unähnliche Punkte in der Visualisierung weiter voneinander entfernt darstellt, erzeugt es eindeutige,
lesbare Cluster, die die zugrunde liegende
Struktur der Trainingsdaten offenlegen. Der Algorithmus
lernt durch unüberwachtes Lernen effektiv die beste Kartendarstellung, indem er die
Divergenz zwischen den hochdimensionalen und niedrigdimensionalen Wahrscheinlichkeitsverteilungen minimiert.
Real-World-Anwendungen in AI
t-SNE ist ein Standardwerkzeug für die
explorative Datenanalyse (EDA) und Modelldiagnostik
. Es ermöglicht Ingenieuren zu „sehen”, was ein Modell lernt.
-
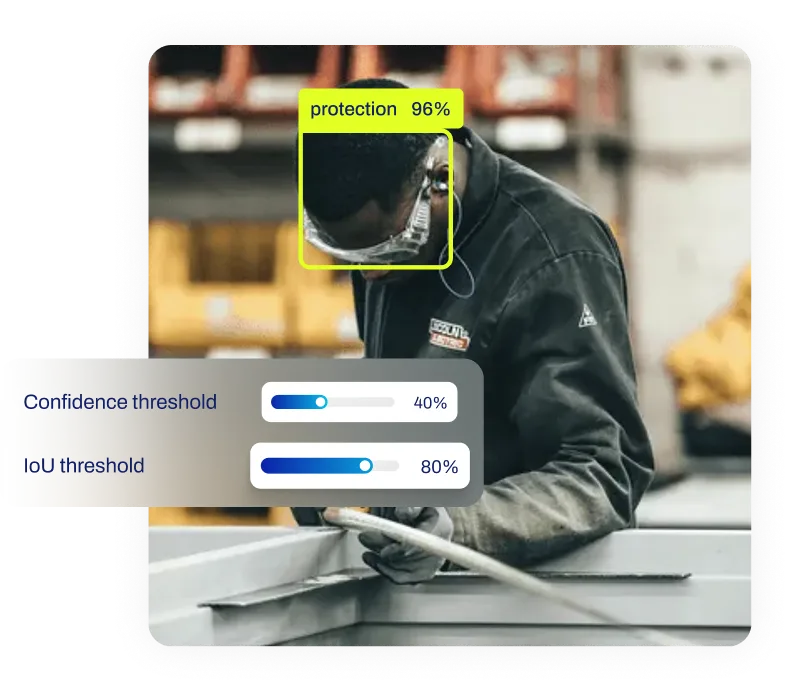
Überprüfen von Computer-Vision-Funktionen: Bei Objektdetektions-Workflows mit Modellen wie
YOLO26 müssen Entwickler häufig überprüfen, ob das Netzwerk
zwischen visuell ähnlichen Klassen unterscheiden kann. Durch Extrahieren der
Feature-Maps aus den letzten Schichten des Netzwerks
und deren Projektion mit t-SNE können Ingenieure visualisieren, ob Bilder von „Katzen” getrennt von
„Hunden” gruppiert werden. Wenn die Cluster gemischt sind, deutet dies darauf hin, dass die
Merkmalsextraktionsfähigkeiten des Modells
verbessert werden müssen.
-
Natürliche Sprachverarbeitung (NLP): t-SNE wird häufig zur Visualisierung von Wort-Embeddings verwendet.
Wenn hochdimensionale Wortvektoren (oft
über 300 Dimensionen) in 2D projiziert werden, gruppieren sich Wörter mit ähnlicher semantischer Bedeutung auf natürliche Weise. Beispielsweise könnte
ein t-SNE-Diagramm einen Cluster mit den Begriffen „König”, „Königin”, „Prinz” und
„Monarch” zeigen, was verdeutlicht, dass das
Modell zur Verarbeitung natürlicher Sprache (Natural Language Processing, NLP)
das Konzept der Königsfamilie erfasst.
-
Genomik und Bioinformatik: Forscher verwenden t-SNE, um Einzelzell-RNA-Sequenzierungsdaten zu visualisieren. Durch die
Reduzierung Tausender Genexpressionswerte auf ein 2D-Diagramm können Wissenschaftler verschiedene Zelltypen identifizieren und
Entwicklungsverläufe nachverfolgen, was zur Entdeckung neuer biologischer Erkenntnisse und Krankheitsmarker beiträgt.
Vergleich mit PCA
Es ist wichtig, t-SNE von der
Hauptkomponentenanalyse (PCA) zu unterscheiden, einer weiteren gängigen Reduktionstechnik.
-
PCA ist eine lineare Technik, die sich auf die Erhaltung der globalen Varianz der Daten konzentriert. Sie ist
deterministisch und rechnerisch effizient, wodurch sie sich hervorragend für die anfängliche Datenkomprimierung oder Rauschunterdrückung eignet.
-
t-SNE ist eine nichtlineare Technik, die sich auf die Erhaltung lokaler Nachbarschaften konzentriert. Sie ist probabilistisch
(stochastisch) und rechenintensiver, erzeugt jedoch weitaus bessere Visualisierungen für komplexe, nichtlineare
Mannigfaltigkeiten.
Eine gängige Vorgehensweise bei der Datenvorverarbeitung besteht darin,
zunächst PCA zu verwenden, um die Daten auf eine überschaubare Größe (z. B. 50 Dimensionen) zu reduzieren, und dann t-SNE für die endgültige
Visualisierung anzuwenden. Dieser hybride Ansatz reduziert die Rechenlast und filtert Störsignale heraus, die das t-SNE-Ergebnis beeinträchtigen könnten
.
Python : Visualisierung von Merkmalen
Das folgende Beispiel zeigt, wie man scikit-learn Anwendung von t-SNE auf einen synthetischen Datensatz. Dieser
Workflow spiegelt wider, wie man aus einem Deep-Learning-Modell extrahierte Merkmale visualisieren könnte.
import matplotlib.pyplot as plt
from sklearn.datasets import make_blobs
from sklearn.manifold import TSNE
# Generate synthetic high-dimensional data (100 samples, 50 features, 3 centers)
X, y = make_blobs(n_samples=100, n_features=50, centers=3, random_state=42)
# Apply t-SNE to reduce dimensions from 50 to 2
# 'perplexity' balances local vs global aspects of the data
tsne = TSNE(n_components=2, perplexity=30, random_state=42)
X_embedded = tsne.fit_transform(X)
# Plot the result to visualize the 3 distinct clusters
plt.scatter(X_embedded[:, 0], X_embedded[:, 1], c=y)
plt.title("t-SNE Projection of High-Dimensional Data")
plt.show()
Wichtige Überlegungen
Obwohl t-SNE leistungsstark ist, erfordert es eine sorgfältige
Hyperparameter-Optimierung. Der
Parameter „Perplexität“ ist entscheidend; er schätzt im Wesentlichen, wie viele nahe Nachbarn jeder Punkt hat. Eine
zu niedrige oder zu hohe Einstellung kann zu irreführenden Visualisierungen führen. Darüber hinaus bewahrt t-SNE globale Abstände nicht gut
– das bedeutet, dass der Abstand zwischen zwei unterschiedlichen Clustern im Diagramm nicht unbedingt ihren physischen
Abstand im ursprünglichen Raum widerspiegelt. Trotz dieser Nuancen bleibt es eine grundlegende Technik zur Validierung von
Computer-Vision-Architekturen (CV) und zum
Verständnis komplexer Datensätze. Benutzer, die große Datensätze verwalten, nutzen häufig Ultralytics , um ihre Daten zu organisieren, bevor sie solche
tiefgreifenden Analysen durchführen.









.webp)